- Nat Commun | 海南大學南繁學院羅杰團隊發布脂肪酸代謝調控水稻產量重磅成果
- 2024年08月09日來源:中國網
提要:大多數生物的表皮都被角質層覆蓋,角質是角質層的主要成分之一,主要由16和18碳的長鏈羥基脂肪酸組成。脂肪酸代謝產物羥基脂肪酸和羥基單酰基甘油(HMG)占角質單體的30%-60%。角質單體的合成涉及長鏈酰基輔酶A合成酶(LACS)、細胞色素P450酶(CYP450)和甘油-3磷酸酰基轉移酶(GPAT),但反應順序不確定,可能因物種而異。

文章導讀:
大多數生物的表皮都被角質層覆蓋,角質是角質層的主要成分之一,主要由16和18碳的長鏈羥基脂肪酸組成。脂肪酸代謝產物羥基脂肪酸和羥基單酰基甘油(HMG)占角質單體的30%-60%。角質單體的合成涉及長鏈酰基輔酶A合成酶(LACS)、細胞色素P450酶(CYP450)和甘油-3磷酸酰基轉移酶(GPAT),但反應順序不確定,可能因物種而異。之前的研究中報道了脂肪酸還原酶(OsFAR2)和OsCYP704B2在水稻花藥角質生物合成中的作用。角質層除了控制水分流失外,還通過影響器官分化影響花器官的發育,從而影響作物產量。轉錄因子如AP2、HD-ZIP和MYB已被證明可調節角質層合成。值得注意的是,雖然MADS家族轉錄因子廣泛調控植物花器官發育,但其對角質層的調控仍未被發現。
2024年8月6日,nature communicaitons在線發表了海南大學羅杰教授團隊利用重測序、廣泛靶向代謝組技術手段,確定了真核脂肪酸和植物初級代謝基因簇(參與合成和運輸)通過調節花藥育性來控制水稻產量。進一步分析了這一初級代謝基因簇的調控及其進化機制,為禾科作物的生態適應和雜交育種提供依據和遺傳資源。

研究摘要:
真核生物基因組中存在種類繁多的代謝基因簇,但脂肪酸代謝基因簇尚未被發現。本研究結合代謝和表型全基因組關聯研究,在3號染色體(FGC3)上發現了一個包含6個基因的脂肪酸代謝基因簇的主要位點,該基因簇控制角質單體羥基單酰基甘油(HMGs)的含量和水稻產量,可能是通過FGC3成員轉錄的變化來實現的。研究發現HMG在內質網中依次由OsFAR2、OsKCS11、OsGPAT6、OsCYP704B2合成,隨后由OsABCG22和OsLTPL82轉運到外質體。FGC3成員突變導致HMG減少,致使雄性生殖發育缺陷和產量顯著下降。OsMADS6和OsMADS17直接調控FGC3,從而影響雄性生殖和產量。FGC3在禾科中是保守的,很可能形成于原禾屬植物Pharus latifolius分化之前。所鑒定的真核脂肪酸和植物初級代謝基因簇對禾本科植物的起源和進化有重要影響,在雜交作物育種中具有應用潛力。
部分研究結果:
1.mGWAS和pGWAS共同定位脂肪酸代謝基因簇
為了評估水稻的脂質變異,從全球533種水稻(Oryza sativa)中收集了糙米樣本,并使用廣泛靶向代謝組學分析策略對脂質代謝物進行了高通量定量分析,共鑒定到267種脂質代謝物。結果表明游離脂肪酸(FFAs)和羥基脂肪酸(HFAs)幾乎都在粳稻品種中富集。代謝全基因組關聯研究(mGWAS)顯示,3號染色體上3.6 Mb的一個主要位點由這些脂肪酸定位(Fig. 1a)。有趣的是,表型全基因組關聯研究(pGWAS)揭示了這個位點與水稻單株產量(圖1a)和小穗育性共定位。進一步分析揭示了脂肪酸含量與單株產量之間的強相關性(圖1b)。結合脂肪酸的代謝途徑和該位點的基因注釋,鑒定出5個主要候選基因(圖1c)。此外在該位點發現了兩個注釋基因。
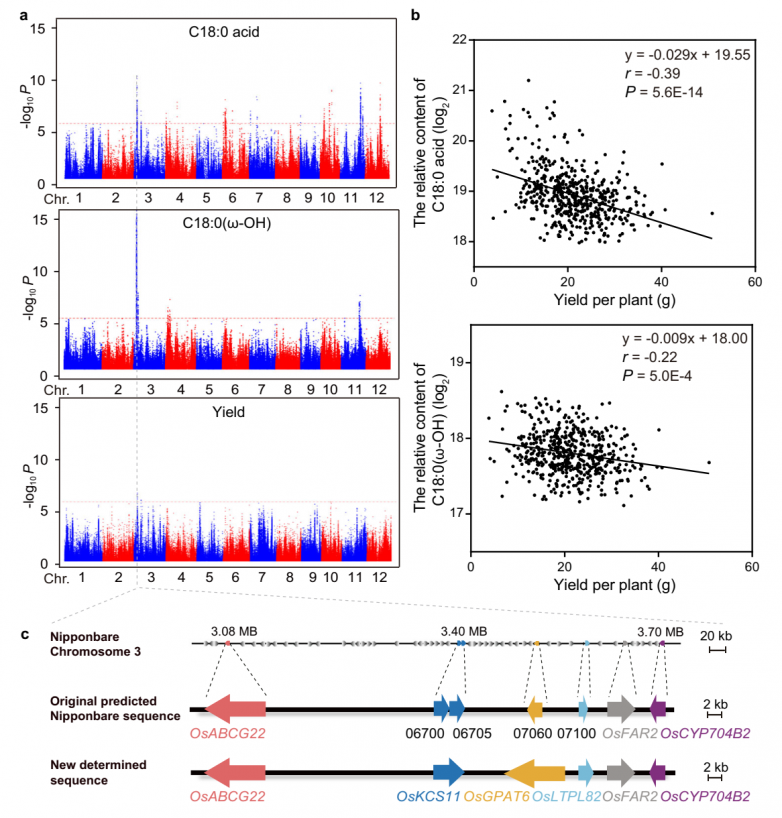
圖1.候選基因篩選分析
2.FGC3合成成員功能分析
為了研究這些合成成員(OsFAR2、OsKCS11、OsGPAT6和OsCYP704B2)的代謝作用,對這些候選基因進行了獨立克隆。亞細胞定位表明,OsFAR2主要位于質體中,而OsKCS11、OsCYP704B2和OsGPAT6位于內質網,分別顯示了它們各自的功能區域。
將OsFAR2重組蛋白在大腸桿菌原核表達系統中表達,并與棕櫚酰酰基載體蛋白(C16:0 ACP)孵育,用標準品鑒定其反應產物為棕櫚醇(圖2a,反應I),當OsFAR2與棕櫚酰輔酶a (C16:0 CoA)孵育時,也檢測到C16:0醇的產生,但比反應I的速率低(圖2a)。以上結果表明,OsFAR2可以在體外催化合成C16:0醇,可用于合成下游代謝反應所需的C16:0。
然后利用酵母表達系統研究了預測的膜定位蛋白OsKCS11、OsCYP704B2和OsGPAT6的體外酶活性。研究結果發現OsKCS11用輔酶A (CoA)激活脂肪酸C16:0,產生C16:0 CoA(經商業標準鑒定;圖2b,反應III)。當羥基脂肪酸C16:0(ω-OH)作為底物時,生成的化合物的質量與M /z 1022.3434處的前體離子[M+H]+相對應(圖2b,反應IV),通過化學合成的標準物鑒定出16羥棕櫚酰基CoA 。這些結果表明OsKCS11可以同時使用脂肪酸和羥基脂肪酸作為底物。進一步實驗發現OsCYP704B2對C16:0和C16:0 CoA都有活性,分別生成C16:0(ω-OH) 和C16:0(ω-OH) CoA(圖2c,反應V和VI)。最后發現OsGPAT6可以與C16:0 CoA和C16:0(ω-OH) CoA反應,并將一個酰基轉移到G3P上,生成2-棕櫚酰甘油(MG16: 0,5)和16羥基十六烷酰甘油(HMG16: 0;圖2d,反應VII和VIII)。這些結果表明OsKCS11、OsCYP704B2和OsGPAT6催化了連續的代謝反應。當這些蛋白與C16:0孵育時,確實是這樣的情況,導致產生C16:0輔酶A作為中間產物,HMG16:0作為終產物(圖2e)。這些結果也提示了HMG生物合成的兩種可能的合成途徑,即OsCYP704B2-OsKCS11OsGPAT6和OsKCS11-OsCYP704B2-OsGPAT6,它們形成了一條發散通路(圖2f)。
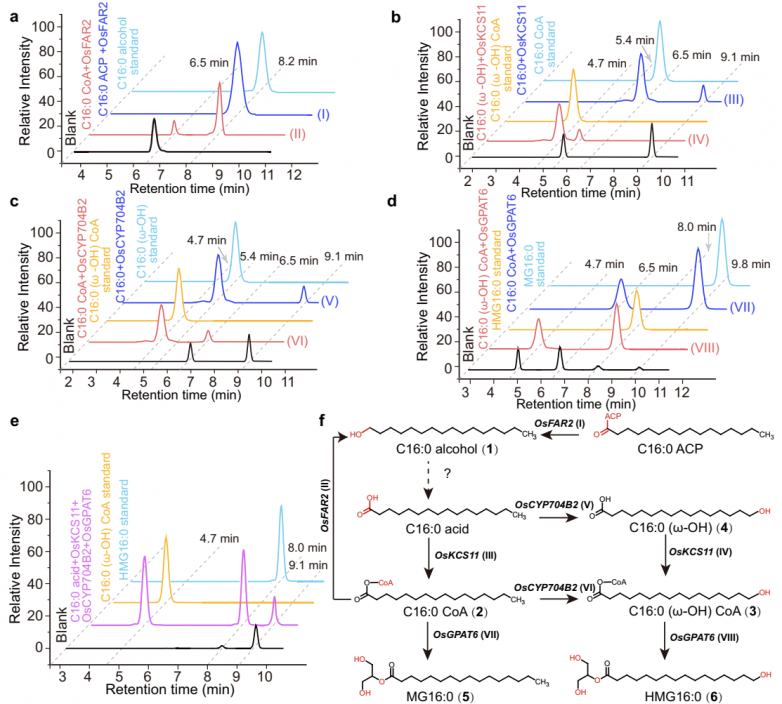
圖2.FGC3的功能分析
研究小結:
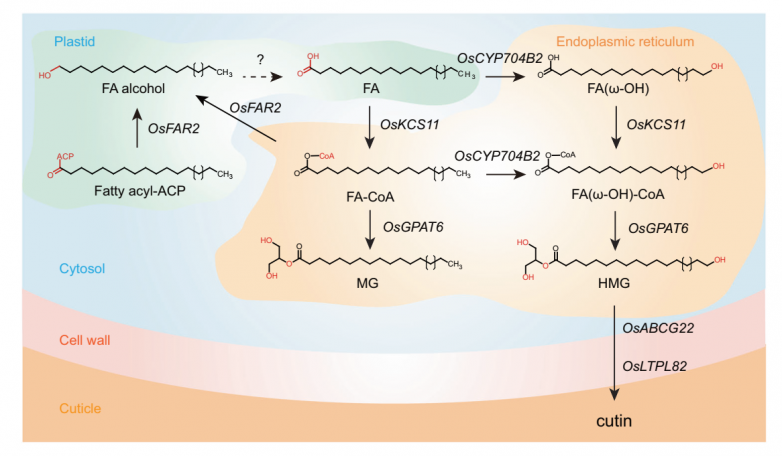
角質素單體生物合成途徑的模型:重新合成和酯化的脂肪酰基ACPs首先被OsFAR2在質體中還原為脂肪醇,然后通過未知基因產生脂肪酸,然后擴散到內質網(ER)。角質素單體HMGs隨后可在內質網中通過OsCYP704B2-OsKCS11-OsGPAT6或OsKCS11OsCYP704B2-OsGPAT6兩種主要合成途徑中的任何一種,通過OsABCG22和OsLTPL82將其轉運到細胞壁的外質體表皮,在此沉積角質素。黑色箭頭表示本文中驗證的步驟,虛線箭頭表示未知步驟。
與國內專家們的研究腳步保持一致,我們也步履不停,在2024年上半年我們已經完成了植物廣泛靶向?代謝組產品的升級,本次升級主要包括3個方面:
1.數據庫升級:本次升級主要增加的為次生代謝物,數據庫目前包含35000+物質,其中33000+種均為次生代謝物;
2.檢出升級:檢出有了顯著的提升,目前植物廣泛靶向?代謝組的最高檢出可以達到4000個物質,Level1平均可以達到600+,最高可到900+;
3.分析升級:MetMapTM的分析已經升級到V2.0版本,包含有60條代謝通路,相較于V1.2版本新增30條通路,通路覆蓋黃酮、萜類、生物堿、香豆素等次生物質通路,升級后的MetMap通路為KEGG擴展了2800+種新物質;
4.工具升級:基于邁維云搭建全新的物質功能詞典V2.0查詢平臺,累計超過3500條物質功能信息,為核心功能物質篩選加速;
歡迎老師們咨詢~